NOVEDADES en CHEMDRAW 25.0 y 25.5 - ¿Las conoces?
- Detalles
- Categoría: Signals ChemDraw
- Visto: 1242
Os traemos las novedades de ChemDraw 25.5, una actualización que llega con mejoras pensadas para pulir aún más el trabajo diario. Pero antes de entrar en materia, merece la pena echar un vistazo rápido a las funcionalidades que incorporó la versión 25 el pasado mes de junio, y que sentaron las bases de esta evolución.
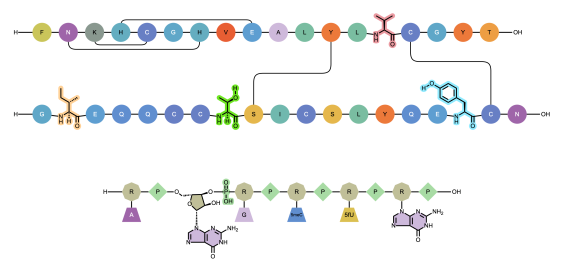
Se han realizado mejoras significativas en la forma en que ChemDraw renderiza secuencias biopoliméricas complejas para proporcionar una representación clara y concisa de las secuencias complejas. Estas mejoras incluyen actualizaciones en la visualización y enrutamiento de enlaces cruzados entre secuencias, así como visualizaciones estructurales limpias para todos los monómeros que pueden expandirse dentro de una secuencia.
Los enlaces cruzados entre secuencias de biopolímeros también están ahora curvados, reemplazando el diseño anterior de esquinas afiladas. Este cambio mejora significativamente la claridad visual de estructuras complejas, permitiendo a los usuarios distinguir fácilmente entre enlaces cruzados y enlaces químicos entre monómeros.
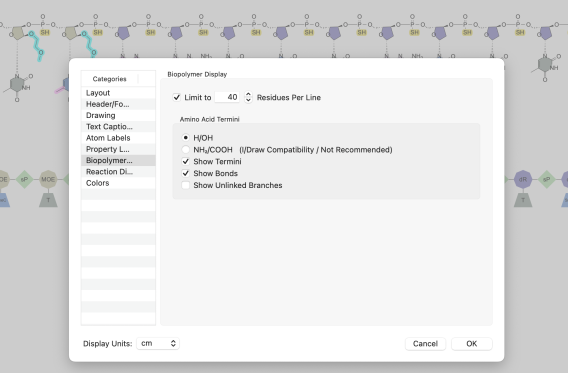
Ahora existen opciones mejoradas para el dibujo y la exhibición de biopolímeros en ChemDraw. Además de poder establecer el número de residuos por línea para biopolímeros, ahora hay una nueva función disponible en la configuración del documento de biopolímeros que permite a los usuarios desactivar por completo el envolvimiento de líneas de secuencias. Cuando esta configuración está desactivada, los biopolímeros se dibujarán en una línea única y continua sin ningún envolvimiento. Por último, la opción de residuos por bloque ha sido eliminada de la configuración de pantalla de biopolímeros.
Los químicos pueden ahora trazar manualmente enlaces de hidrógeno entre cadenas complementarias de ADN/ARN que se generan en ChemDraw que contienen bases nitrogenadas no naturales. La herramienta de enlace de hidrógeno puede seleccionarse y utilizarse para colocar los enlaces de hidrógeno apropiados según el emparejamiento de bases Watson-Crick del análogo natural de bases nitrogenadas.
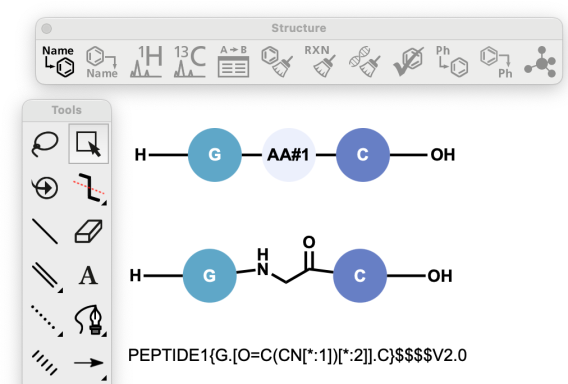
Los SMILES mapeados por atom ahora se soportan en ChemDraw al insertar cadenas de HELM o pegarlas al lienzo. Además, al generar una cadena HELM para una secuencia que incluye un monómero desconocido o en línea, la cadena HELM resultante usará ahora SMILES mapeados por átomos. Esto reemplaza el formato extendido SMILES que se usaba anteriormente.
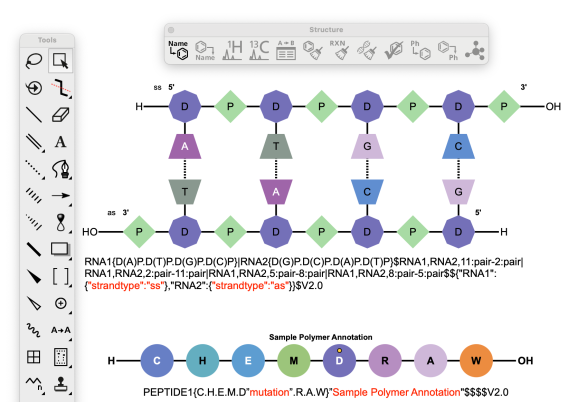
En ChemDraw se soportan anotaciones en línea de monómeros y polímeros, así como anotaciones sense/antisense y designaciones 5'/3', que aparecen cuando están presentes en un cdxml o forman parte de una cadena HELM pegada. También persisten ahora en archivos MOL en SGROUPS.
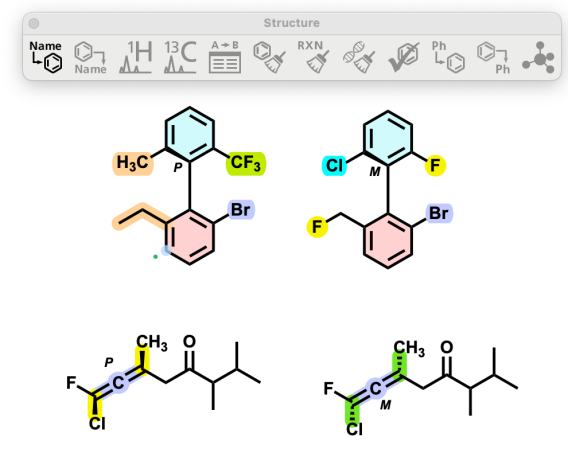
Los estereocentros de alenos y atropisomeros ahora están etiquetados con M y P (en lugar de los antiguos R y S).
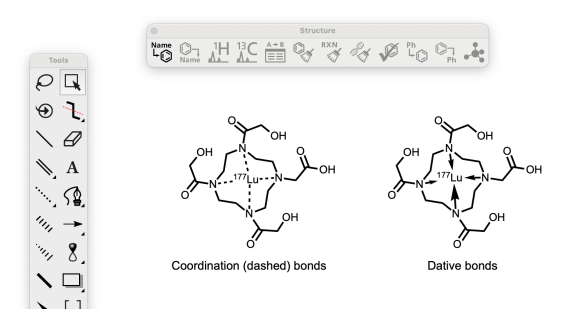
ChemDraw ahora trata los enlaces de coordinación y dativos de forma idéntica, asegurando cálculos de valencia consistentes y percepción estereoquímica para estos tipos de enlaces químicamente equivalentes. Además, los enlaces de coordinación y dativo coinciden entre sí en las búsquedas, pero no coinciden con los enlaces simples.
La gestión de la opción DISPLAY en los archivos MOL ha cambiado, de la siguiente manera:
La instalación de ChemDraw se ha simplificado con esta versión, combinando todas las opciones de usuario/usuario actual y mejorando el comportamiento de los instaladores. Esta actualización simplifica el proceso de instalación al fusionar las opciones de usuario y mejorar el comportamiento general del instalador, haciéndolo más fácil de usar y eficiente.
ChemDraw ya está disponible para Macs ARM e Intel. El tipo de instalación se selecciona en tiempo de ejecución dependiendo de la arquitectura de la máquina.
Ahora hay dos instaladores separados disponibles: uno para ChemDraw y otro para aplicaciones ChemDraw. El instalador de aplicaciones de ChemDraw incluye ChemDraw para Excel, Chem3D, ChemFinder, ChemScript y ChemFinder para Office.
Las mejoras de la versión 25.5 se centran en cálculos de propiedades, manejo de HELM y soporte extendido de Python para ChemScript. Por último, también se han corregido un pequeño número de errores.
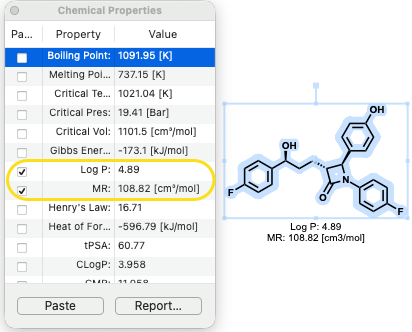
Los cálculos de LogP y Refractividad Molar (RM) ahora se basan en RDKit, ofreciendo resultados más precisos y consistentes en la ventana de Propiedades Químicas.
Los cálculos de propiedades también se han optimizado para mayor eficiencia: al editar estructuras grandes con la ventana de Propiedades Químicas abierta, ahora se ponen actualizaciones en cola para minimizar el retardo y garantizar una experiencia de edición más fluida.
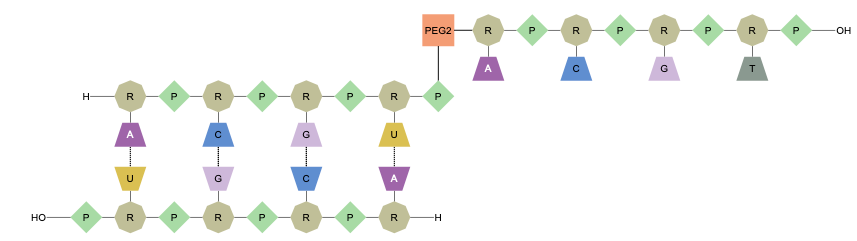
La limpieza de biopolímeros ha sido significativa. Las secuencias añadidas a los hilos complementarios ahora están correctamente dispuestas y las secuencias colgantes se ordenan para mayor claridad.
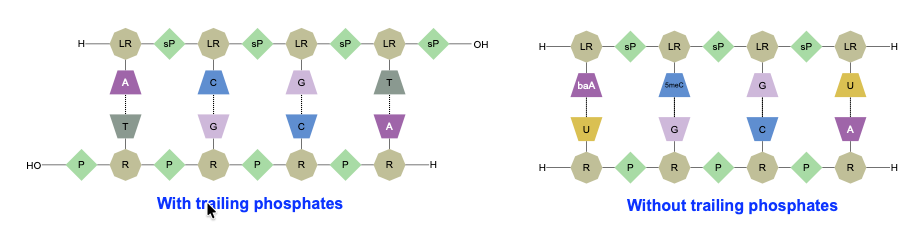
Los fosfatos delanteros y posteriores, cuando se omiten de una secuencia, se emparejan con fosfatos naturales en la cadena complementaria.
El comportamiento de selección de secuencias también ha mejorado: las acciones de doble y triple clic ahora proporcionan una selección intuitiva de monómeros, secuencias y grupos.
Finalmente, la opción del menú Editar > Copiar como HELM (Analógico Natural) ha sido renombrada a Editar > Copiar como FASTA para mayor claridad.
ChemScript ahora soporta Python las versiones 3.13.x, 3.10.x y 3.9.x, lo que ofrece a los desarrolladores mayor flexibilidad para la integración y la automatización.
Chemdraw 25.0
Mejoras avanzadas en biopolímeros (HELM)

Se han realizado mejoras significativas en la forma en que ChemDraw renderiza secuencias biopoliméricas complejas para proporcionar una representación clara y concisa de las secuencias complejas. Estas mejoras incluyen actualizaciones en la visualización y enrutamiento de enlaces cruzados entre secuencias, así como visualizaciones estructurales limpias para todos los monómeros que pueden expandirse dentro de una secuencia.
Los enlaces cruzados entre secuencias de biopolímeros también están ahora curvados, reemplazando el diseño anterior de esquinas afiladas. Este cambio mejora significativamente la claridad visual de estructuras complejas, permitiendo a los usuarios distinguir fácilmente entre enlaces cruzados y enlaces químicos entre monómeros.

Ahora existen opciones mejoradas para el dibujo y la exhibición de biopolímeros en ChemDraw. Además de poder establecer el número de residuos por línea para biopolímeros, ahora hay una nueva función disponible en la configuración del documento de biopolímeros que permite a los usuarios desactivar por completo el envolvimiento de líneas de secuencias. Cuando esta configuración está desactivada, los biopolímeros se dibujarán en una línea única y continua sin ningún envolvimiento. Por último, la opción de residuos por bloque ha sido eliminada de la configuración de pantalla de biopolímeros.
Los químicos pueden ahora trazar manualmente enlaces de hidrógeno entre cadenas complementarias de ADN/ARN que se generan en ChemDraw que contienen bases nitrogenadas no naturales. La herramienta de enlace de hidrógeno puede seleccionarse y utilizarse para colocar los enlaces de hidrógeno apropiados según el emparejamiento de bases Watson-Crick del análogo natural de bases nitrogenadas.

Los SMILES mapeados por atom ahora se soportan en ChemDraw al insertar cadenas de HELM o pegarlas al lienzo. Además, al generar una cadena HELM para una secuencia que incluye un monómero desconocido o en línea, la cadena HELM resultante usará ahora SMILES mapeados por átomos. Esto reemplaza el formato extendido SMILES que se usaba anteriormente.

En ChemDraw se soportan anotaciones en línea de monómeros y polímeros, así como anotaciones sense/antisense y designaciones 5'/3', que aparecen cuando están presentes en un cdxml o forman parte de una cadena HELM pegada. También persisten ahora en archivos MOL en SGROUPS.
Inteligencia química mejorada

Los estereocentros de alenos y atropisomeros ahora están etiquetados con M y P (en lugar de los antiguos R y S).

ChemDraw ahora trata los enlaces de coordinación y dativos de forma idéntica, asegurando cálculos de valencia consistentes y percepción estereoquímica para estos tipos de enlaces químicamente equivalentes. Además, los enlaces de coordinación y dativo coinciden entre sí en las búsquedas, pero no coinciden con los enlaces simples.
La gestión de la opción DISPLAY en los archivos MOL ha cambiado, de la siguiente manera:
- El valor de DISP se establece en COORD por defecto si no está presente en el archivo MOL
- El DISP siempre está configurado en COORD para enlaces de coordinación
Instalación y mejoras del sistema
La instalación de ChemDraw se ha simplificado con esta versión, combinando todas las opciones de usuario/usuario actual y mejorando el comportamiento de los instaladores. Esta actualización simplifica el proceso de instalación al fusionar las opciones de usuario y mejorar el comportamiento general del instalador, haciéndolo más fácil de usar y eficiente.
ChemDraw ya está disponible para Macs ARM e Intel. El tipo de instalación se selecciona en tiempo de ejecución dependiendo de la arquitectura de la máquina.
Ahora hay dos instaladores separados disponibles: uno para ChemDraw y otro para aplicaciones ChemDraw. El instalador de aplicaciones de ChemDraw incluye ChemDraw para Excel, Chem3D, ChemFinder, ChemScript y ChemFinder para Office.
ChemDraw 25.5
Las mejoras de la versión 25.5 se centran en cálculos de propiedades, manejo de HELM y soporte extendido de Python para ChemScript. Por último, también se han corregido un pequeño número de errores.
Cálculo de propiedades

Los cálculos de LogP y Refractividad Molar (RM) ahora se basan en RDKit, ofreciendo resultados más precisos y consistentes en la ventana de Propiedades Químicas.
Los cálculos de propiedades también se han optimizado para mayor eficiencia: al editar estructuras grandes con la ventana de Propiedades Químicas abierta, ahora se ponen actualizaciones en cola para minimizar el retardo y garantizar una experiencia de edición más fluida.
Mejoras en HELM

La limpieza de biopolímeros ha sido significativa. Las secuencias añadidas a los hilos complementarios ahora están correctamente dispuestas y las secuencias colgantes se ordenan para mayor claridad.

Los fosfatos delanteros y posteriores, cuando se omiten de una secuencia, se emparejan con fosfatos naturales en la cadena complementaria.
El comportamiento de selección de secuencias también ha mejorado: las acciones de doble y triple clic ahora proporcionan una selección intuitiva de monómeros, secuencias y grupos.
Finalmente, la opción del menú Editar > Copiar como HELM (Analógico Natural) ha sido renombrada a Editar > Copiar como FASTA para mayor claridad.
Soporte en Python
ChemScript ahora soporta Python las versiones 3.13.x, 3.10.x y 3.9.x, lo que ofrece a los desarrolladores mayor flexibilidad para la integración y la automatización.

