Chem3D junto interfaz GAMESS
- Detalles
- Categoría: ChemOffice
- Visto: 35090
Este software de última generación está diseñado para el modelado molecular de estructuras químicas o biológicas de cualquier tamaño. De todas las ciencias naturales, el programa está específicamente recomendado para investigadores químicos y biológicos a cualquier nivel. Los complejos ligando-proteína 3D y las estructuras de ADN se pueden visualizar utilizando gráficos GL abiertos y hardware estéreo. Los enlaces de hidrógeno y las superficies parciales se pueden mostrar y analizar. El software ayuda a construir moléculas pequeñas utilizando la interfaz ChemDraw, al mismo tiempo que muestra la estructura en 3D. También facilita de manera efectiva los cálculos de modelado molecular básico como la alineación, muestreo conformacional estocástico, comprobador diedro, experimentos MM2 y mecánica cuántica molecular MMFF94. Este software es muy recomendable para realizar cálculos ab initio y semi-empíricos.
Los comandos de esta interfaz son muy fáciles de usar, ayudan a analizar una amplia y diversa gama de moléculas, incluso biológicas y nanomoléculas. Además de los elementos de menú estándar como “Archivo”, “Editar”, “Ver”, “Ventana” y “Ayuda”, el programa proporciona opciones de “Estructura”, “Cálculos”, “Superficie” y “En línea”. El usuario encontrará un “cursor” que permite dibujar, borrar, rotar y/o modificar cualquier molécula simplemente arrastrando el mismo. Este software permite además modificar el color del fondo y las sombras de las estructuras dibujadas.
El menú “Archivo” incluye más de veinte ejemplos de estructuras de todo tipo. Las estructuras dibujadas por el usuario pueden ser guardadas en formato imagen (MDL, MOPAC, JPG, TIF, etc). Las opciones de copiado presentes en el menú “Editar” no solo permiten pegar las estructuras dibujadas en otros documentos, sino que también ofrecen la flexibilidad de convertir a otros formatos; ChemDraw, SMILES e imágenes, por ejemplo.
En el apartado “Ver”, la visualización de la estructura puede ajustarse de varias formas, podemos mostrar los átomos de hidrógeno y los pares de electrones no enlazantes. El menú “Estructura” ayuda a calcular las longitudes de enlace, los ángulos estándar y ángulos diedros.
La herramienta analítica más importante en este software es el menú “Cálculos”, que permite utilizar el método Hückel extendido para calcular cargas y superficies, seguido del uso de MM2 que permite realizar cálculos de optimización energética, dinámica molecular y cálculo de propiedades para moléculas pequeñas.
La interfaz nos ofrece además la posibilidad de realizar cálculos vía MMFF94, mediante minimización de energía, dinámica molecular y gradientes.
MMFF94 para cálculos de dinámica molecular
En versiones anteriores la función MMFF94 ya era una herramienta poderosa para el cálculo de energías mínimas, pero gracias a la mejora en la versión 12.0 está equipado para realizar cálculos de dinámica molecular, estos cálculos son la herramienta principal en el estudio teórico de moléculas biológicas de gran tamaño.
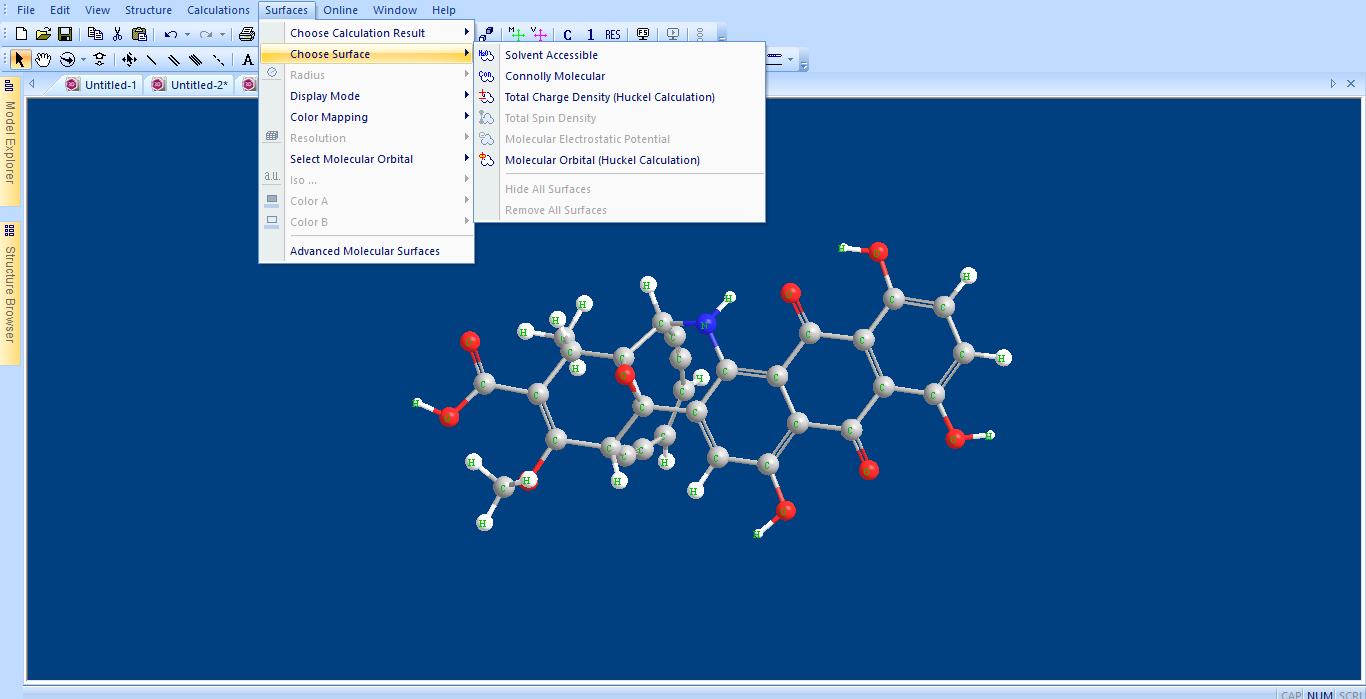
Cálculo de superficies
Esta opción le permite visualizar las superficies moleculares. En la mayoría de los casos será preciso que el usuario realice un cálculo extendido de Hückel, MOPAC o Gaussiano antes de obtener información sobre la superficie molecular.
Barra de herramientas de cálculo
Proporciona un icono de escritorio para realizar minimización energética vía MM2. También proporciona al usuario un comando de detección e indicador de estado del cálculo realizado.
MMFF94
Puede utilizarse para realizar cálculos de minimización de energía para proteínas u otras estructuras biológicas complejas.
Los cálculos de campos de fuerza mediante modelado molecular pueden consumir mucho tiempo y ser poco prácticos para moléculas de gran tamaño. Este problema puede solventarse utilizando multiprocesadores. Por ejemplo, con dos procesadores funcionando en paralelo, el cálculo se realizará dos veces más rápido.
Cálculo de energía potencial
El usuario puede realizar un cálculo MMFF94 para obtener la energía potencial del modelo de investigación, incluso sin haber realizado previamente un cálculo de minimización de energía.
Cálculos electrostáticos
La energía electrostática es una función que depende de los átomos enlazados, su distancia interatómica y una expresión dieléctrica molecular que da cuenta de la atenuación de la interacción electrostática con el medio ambiente. Se trata de interacciones entre partículas o átomos que están muy cerca o muy alejados. Se aproxima a las interacciones electrostáticas completas y, por lo tanto, no se requiere ningún método de corte.
Cálculos de Van der Waals
Este tipo de fuerzas aparecen a corto alcance y pierden intensidad con la distancia. Las repulsiones ocurren cuando la distancia entre átomos que interactúan es ligeramente menor que la suma de la distancia de sus radios de contacto. El cálculo de Van der Waals es un cálculo de energía no ligada. A medida que aumenta el número de átomos, los cálculos de enegía de Van der Waals pueden suponer largos periodos de tiempo. Se le mostrará al usuario una advertencia si se produce un error.
Minimización energética
Una de las aplicaciones más comunes para cualquiera de los métodos anteriormente nombrados es realizar cálculos de minimización de energía. Al constituir un modelo, la ubicación asignada a cada átomo puede no representar con precisión su ubicación en la molécula real. El modelo especificado puede representar una cepa de alta energía en varios enlaces o deformación conformacional entre átomos, por lo que no representa con precisión la molécula real. Para garantizar que el modelo sea específicamente como la estructura real, se debe realizar un cálculo de minimización de energía MM2 o MMFF94. Al realizar cualquiera de los dos cálculos, Chem3D examina el modelo e identifica los distintos tipos de átomos. A continuación, calcula una nueva posición para cada átomo con el fin de minimizar la energía potencial acumulativa del modelo molecular.
Cálculos múltiples
Este software ofrece la opción de minimizar varios modelos simultáneamente. Sin embargo, al comenzar a minimizar un segundo modelo, si un cálculo está en proceso, el primero se retrasa hasta que el último se detiene.
Chem3D puede realizar cualquier acción que no mueva, agregue o elimine una parte del modelo especificado. Es decir, el usuario puede mover ventanas durante la minimización de energía, cambiar la configuración o escalar el modelo especificado.
Dinámica molecular
Este proceso usa la mecánica newtoniana para simular el movimiento de los átomos, así como para sumar o restar energía cinética a medida que la temperatura de las moléculas aumenta o disminuye. Permite al usuario acceder al espacio conformacional disponible para un modelo. El proceso se realiza almacenando iteraciones de la ejecución de la dinámica molecular para examinarlo con posterioridad. La simulación de una dinámica molecular se puede realizar utilizando cualquiera de los campos de fuerza que Chem3D ofrece, MM2 o MMFF94.
Propiedades químicas
Gracias a este método el usuario puede calcular la energía estérica total para la conformación actual del modelo (el marco activo, si hay más de uno).
GAMESS: Descripción general
El sistema general de estructura electrónica atómica y molecular (GAMESS) es un paquete de química cuántica ab initio completo. Calcula las funciones de onda utilizando RHF, ROHF, UHF, GVB y MCSCF. Además, las correcciones de energía CI y MP2 están disponibles para algunas de las funciones calculadas.
GAMESS es una aplicación de línea de comandos, la información que el usuario debe introducir será únicamente basada en texto. Chem3D sirve como interfaz gráfica de usuario (GUI) frontal, que permite crear y ejecutar trabajos GAMESS desde dentro de la aplicación. Aunque la aplicación GAMESS se instale automáticamente con Chem3D, el usuario debe aceptar un acuerdo de licencia y registrar el software antes de comenzar a usarlo. Chem3D realiza esta función de manera automática tan pronto como se ejecuta GAMESS en el servidor. Las opciones de cálculo en el menú de la interfaz GAMESS incluyen minimización, estado de transición, frecuencia, espectro IR, Raman y cálculos de espectros de RMN para isótopos de protón y carbono.
Cuando se selecciona una de las opciones aparece el cuadro de diálogo de la interfaz GAMESS, mostrándose los parámetros predeterminados que podrán modificarse posteriormente. En las formulaciones ab initio los cálculos pueden realizarse por métodos de HF con amplia gama de conjuntos de bases (mini, midi, ST 0-2G, ST 0-3G a 6G, 3-21 G, 6-21 G, 4-31 G, 5-31 G, 6-31 G, 6-311 G, D2V, DHT2V, MC, SBKJC y HW). Otros métodos para formulaciones ab initio son MP2, DFT, GVB y MCSCF, así como los métodos agrupados acoplados. La configuración de la interfaz incluye: advance1, advance 2 y general. Las propiedades que se pueden calcular son: Densidad electrónica de dipolo, potencial electrostático, energía cinética, superficies moleculares, energía potencial y total, entre otros. Para los cálculos de superficie existe un menú adicional que puede calcular accesibilidad del disolvente, densidad de cambio y orbitales moleculares.
Conclusión
En su clasificación general, este software proporciona muchos beneficios a los usuarios que participen en el aprendizaje superior formal e informal, así como a los docentes en múltiples campos. Es recomendable su uso incluso a niveles de pregrado donde el usuario se encuentra en las primeras etapas de aprendizaje. Chem3D proporciona valores reales de información física y química. Gracias a GAMESS, que puede servir de ayuda al usuario para realizar cálculos cuánticos semi-empíricos y ab initio, los resultados experimentales para, prácticamente, todo tipo de moléculas serán de gran calidad.

